SCALE - Exzellenzcluster

Der Exzellenzcluster
Vom Einzelmolekül zur Zellfunktion
Das SCALE-Konsortium vereint technologische Kompetenzen aus Zellbiologie, Biophysik, Molekularbiologie, Neurobiologie, Chemie, Bioinformatik und Mathematik. Sie entwickeln radikal neue experimentelle Techniken, um das Innere von Zellen abzubilden, zu simulieren und ihr Verhalten vorherzusagen. Diese Forschungsarbeiten liefern wichtige neue Erkenntnisse über bakterielle Resistenz, Entzündungen, neurodegenerative Erkrankungen und Immunabwehr.
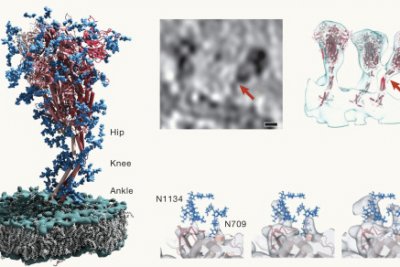
Der menschliche Körper besteht aus Billionen funktionell spezialisierter Zellen. Jede Zelle ist dicht gepackt mit Molekülen, die biochemische Reaktionen durchführen, Energie umwandeln, Signale übertragen oder die Zellen in die Lage versetzen, sich bei Bedarf zu bewegen. Um diese erstaunlich vielfältigen Funktionen ausführen zu können, sind die Zellen in spezialisierte Kompartimente unterteilt, in denen bestimmte Moleküle lokal konzentriert sind. Diese subzelluläre Architektur entsteht dynamisch und wird ständig umgestaltet, um sich den zellulären Bedürfnissen und den Umweltbedingungen anzupassen. Die Moleküle organisieren sich durch synergistische Effekte in einer komplexe Choreographie selbst.
Wissenschaftler:innen haben die Hauptakteure der subzellulären Architektur identifiziert und charakterisiert, z. B. die Moleküle, die die zellulären Kraftwerke (Mitochondrien) bilden, oder die Moleküle, die für die Verteidigung gegen bakterielle Infektionen zuständig sind. Wie jedoch die einzelnen Bausteine der subzellulären Architektur in der komplexen Umgebung einer Zelle zusammenwirken, ist nach wie vor unklar. Wir verstehen noch nicht einmal die grundlegenden Konzepte, z. B. wie sich die Zellmembranen zu der komplizierten, vielschichtigen Architektur der Mitochondrien falten oder wie sie eindringende Bakterien umgeben und isolieren. Es ist unmöglich vorherzusagen, wie die subzelluläre Architektur umgestaltet wird, etwa bei Stress oder Krankheit. Bisher war es schwierig, einzelne Moleküle im Inneren von Zellen in hoher räumlicher und zeitlicher Auflösung zu beobachten.
Der SCALE-Cluster verfügt über Kenntnisse in der fortgeschrittenen experimentellen Analyse von Zellen, der computergestützten Modellierung und Simulation von Molekülen und der Untersuchung der subzellulären Organisation, z. B. der Energieumwandlung in Mitochondrien oder der bakteriellen Resistenz. SCALE baut auf diesem Fachwissen auf, um Methoden zur Visualisierung der Bausteine von Zellen bei der Arbeit zu entwickeln und die rechnerische Integration von wissenschaftlichen Daten zu erleichtern. Wir erstellen digitale Zwillinge von Zellen, die bestimmte Zellsegmente bis ins molekulare Detail und in vier Dimensionen simulieren, um sowohl die wissenschaftliche Konzeptualisierung als auch die gesellschaftliche Bildung zu unterstützen. Die digitalen Zwillinge werden zelluläres Verhalten in silico vorhersagen und unser Verständnis z. B. dafür verbessern, wie die mitochondriale Architektur während menschlicher Krankheiten oder der Alterung umgestaltet wird. Letztendlich sollen sie dazu beitragen, neue architektonische Elemente auf der Grundlage von Ansätzen der synthetischen Biologie zu entwerfen.
Im Internal Board ist FIAS-Senior-Fellow Gerhard Hummer vertreten, zu den PIs zählt FIAS-Fellow Roberto Covino, assoziierte Mitglieder sind die FIAS-Fellows Franziska Matthäus und Sebastian Thallmair.
Projektseite
Projektleitung
Sprecherteam
Michaela Müller-McNicoll
Inga Hänelt
Martin Beck
scale-office_at_uni-frankfurt.de
Partner